哺乳動物のセラミド関連脂質生合成
2023.10.28
山地 俊之*
*, 国立感染症研究所 細胞化学部
1.はじめに
スフィンゴ脂質は、スフィンゴシンをはじめとする長鎖塩基(スフィンゴイド塩基とも呼ばれる)を含んだ脂質群の総称であり、細胞の生存に必須の脂質である(1–3)。スフィンゴシンのアミノ基に脂肪酸がアミド結合で付加した構造がセラミド(図1)であり、またスフィンゴシンのC1位の水酸基にリン酸の付加した構造がスフィンゴシン-1-リン酸である。これらスフィンゴシン、セラミド、スフィンゴシン-1-リン酸は代謝中間体としてのみならず、様々な細胞内シグナル分子として機能する。さらにセラミドのC1位の水酸基にホスホコリン、糖鎖、リン酸が付加することで、それぞれスフィンゴミエリンやスフィンゴ糖脂質、セラミド-1-リン酸が生成される。スフィンゴミエリンとスフィンゴ糖脂質は形質膜におけるスフィンゴ脂質の主要な成分であり、コレステロールと共にシグナル伝達のハブとなる脂質マイクロドメイン(脂質ラフト)を形成する。スフィンゴ脂質の機能は多岐にわたっており、本冊子の各章、あるいは他の総説を参考にしていただきたい。本稿では、スフィンゴ脂質の特にセラミドを中心とした比較的単純な構造のスフィンゴ脂質生合成について、酵素の細胞内局在や基質である脂質の輸送に焦点を絞り概説する(図2を参照)。なおセラミドの分解によって生成されるスフィンゴシンやスフィンゴシン-1-リン酸の生合成については本研究会サイトの総説『スフィンゴ脂質恒常性維持のためのセラミド分解経路』を参照していただきたい。
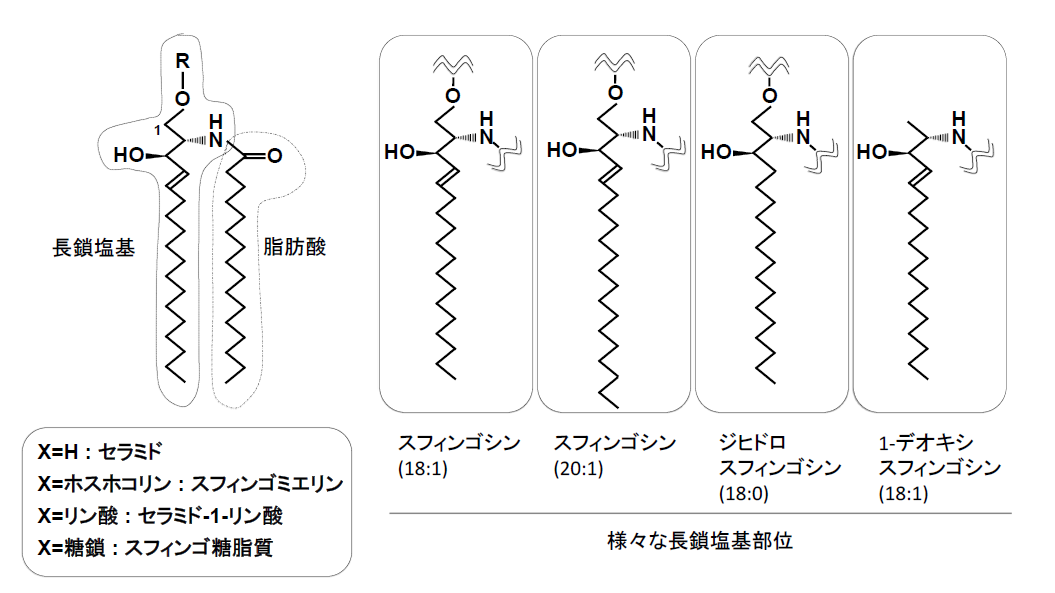
図1 セラミド及び様々な長鎖塩基の構造
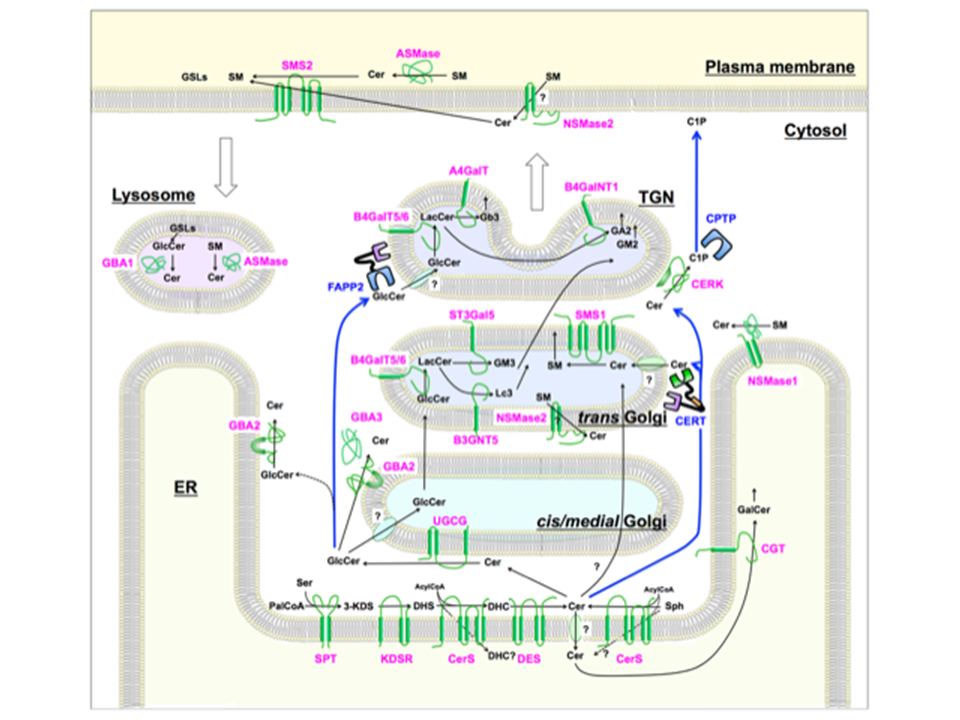
図2 スフィンゴ脂質生合成経路
2.小胞体におけるスフィンゴ脂質生合成–セラミド生合成まで
セリンパルミトイル転移酵素(SPT)
スフィンゴ脂質のde novoの生合成は、アミノ酸のL-セリンと脂肪酸のパルミトイルCoAの縮合反応により、3-ケトジヒドロスフィンゴシン(3-ケトスフィンガニン)を生成するところから始まる(4)。この反応を司るのはセリンパルミトイル転移酵素(SPT)である。SPTはSPTLC1(LCB1)とSPTLC2(LCB2)の2種類のサブユニットによるヘテロ二量体だと考えられていたが(5–8)、近年新たな構成成分として、SPTLC2に似た構造を有するSPTLC3がSPTLC2と同様にSPTLC1とヘテロ二量体を形成すること(9)、また分子量の小さなタンパクであるSPTSSA及びSPTSSBが上記のヘテロ二量体とそれぞれ結合し、新たなサブユニットとして機能することが明らかとなった(10)。すなわち4種類のSPT複合体(SPTLC1/SPTLC2/SPTSSA、SPTLC1/SPTLC3/SPTSSA、SPTLC1/SPTLC2/SPTSSB、SPTLC1/SPTLC3/SPTSSB)が発現していると考えられる。興味深いことに、SPT複合体が作用する脂肪酸の基質特異性はそれぞれ異なり、その結果様々な長さのスフィンゴ塩基が生合成される。SPTSSAはパルミトイルCoA(C16)をよい基質とするため典型的なC18-スフィンゴシンを生成するが、SPTSSBはパルミトイルCoAの他、ステアロイルCoA(C18)を基質とするため、C18-スフィンゴシンに加えC20-スフィンゴシンを生成する(10)。またSPTLC3が複合体に入ると脂肪酸の長さに対する基質認識が甘くなるため、SPTLC1/SPTLC3/SPTSSAの場合パルミトイルCoA より短いミリストイルCoA(C14)も使用可能となり、その結果C16-スフィンゴシンが生成される(11)。またSPTLC1/SPTLC3/SPTSSBの場合も、C16-スフィンゴシンからC-22スフィンゴシンに至る幅広い長鎖塩基が生成される。実際にはSPTSSBは組織特異性があり、またパルミチン酸はミリスチン酸より細胞内で多く含有しているため、ユビキタスに発現している主な長鎖塩基はC18-スフィンゴシンである(10)。一方SPTLC1の点突然変異により、アミノ酸に対する基質特異性が変化し、セリンのみならずアラニンを基質とする特殊な長鎖塩基、1-デオキシスフィンゴシンが生成される(図1)(12)。この1-デオキシスフィンゴシンはセリンの生合成を抑制した場合にも見られ、親水部を形成する水酸基を持たないことから、スフィンゴミエリンやスフィンゴ糖脂質に代謝されず、その結果蓄積することで細胞に対する毒性を示す(13)。なおSPTは小胞体に局在し、活性部位は基質が存在している細胞質側に面している(14,15)。
3-ケトジヒドロスフィンゴシン還元酵素(KDSR)
SPTにより生成された3-ケトジヒドロスフィンゴシンは3-ケトジヒドロスフィンゴシン還元酵素(KDSR/FVT1)により、C3位のカルボニル基がNADPH依存的に還元され、その結果ジヒドロスフィンゴシン(スフィンガニン)が生成される(16,17)。この酵素もSPTと同様小胞体に局在し、活性部位は基質が存在している細胞質側に面している。
セラミド合成酵素(CERSs)
ジヒドロスフィンゴシンのアミド基に様々な長さの脂肪酸(アシルCoA)がアミド結合で付加することにより、ジヒドロセラミドが生成される。この反応を司るのは、哺乳動物では6種類存在するセラミド合成酵素(CerS1-6)である(18,19)。上記のde novoによるジヒドロセラミド生合成の他、salvage経路、すなわちスフィンゴミエリンやスフィンゴ糖脂質の分解によって生じるスフィンゴシンのアシル化によるセラミド生合成もこれらの酵素が担う。それぞれのCerSは異なる鎖長のアシルCoAを基質として(ジヒドロ)セラミドを合成する。例えば、CerS2は主にC20-C26極長鎖脂肪酸を基質とするのに対し、CerS5はC16長鎖脂肪酸を主な基質とする(20–24)。CERSsは主に小胞体に局在しており、CerSの細胞質側が活性に必要であることはプロテアーゼプロテクション法により示されているが(25)、機能的に必須なアミノ酸残基は小胞体の細胞質、内腔の両側に存在している(23)。最近CerSの基質特異性に重要な領域が同定され、小胞体の内腔側に存在する膜貫通領域間ループがアシルCoAの特異性に大きく影響することが明らかになった(26)。よってCerSsがどのように基質であるスフィンゴ塩基やアシルCoAを認識するのか、あるいは小胞体膜のどちら側でセラミド(ジヒドロセラミド)が生合成されるかは議論の余地があり、今後の構造解析の結果が待たれる。
ジヒドロセラミドΔ4-不飽和化酵素(DES1, DES2)
ジヒドロセラミドは、スフィンゴ脂質Δ4-不飽和化酵素DES1によりスフィンゴ骨格のC4、5位(Δ4)がtransに不飽和化されることでセラミドに変換されるか、もしくはDES1と相同性の高いスフィンゴ脂質C4水酸化酵素DES2によりスフィンゴ骨格のC4位が水酸化されることでフィトセラミドに変換される(27,28)。ちなみにDESの名前はもともとDrosophila melanogaster degenerative spermatocyte遺伝子の略として命名された(29)。DES1は小胞体に局在し、N末端のグリシンがミリストイル化されている(30)。またDES1の活性部位は細胞質側に存在すると考えられており、またこの酸化還元反応に必要なシトクロムb5も膜の細胞質側に活性部位を持っている(31–33)。DES2も同様、膜の細胞質側に活性部位を有する(34)。
3.様々な極性基を有するスフィンゴ脂質の生合成
小胞体で生合成されたセラミドは脱アシル化によりスフィンゴシン、続いてスフィンゴシン-1-リン酸が生合成される一方、セラミドの頭部の水酸基に様々な分子が付加することで、スフィンゴミエリン、グルコシルセラミド(GlcCer)、ガラクトシルセラミド(GalCer)、セラミド-1-リン酸(C1P)が生成される。これらセラミド骨格を有したスフィンゴ脂質の多くはゴルジ体で生成される。
スフィンゴミエリン合成酵素
スフィンゴミエリンはホスファチジルコリンのホスホコリンがセラミドに転移されることで産生され(35)、2種類のスフィンゴミエリン合成酵素、SMS1及びSMS2が、この反応を司る(36,37)。電子顕微鏡解析によりSMS1は主にトランスゴルジ槽に局在しており、de novoのスフィンゴミエリン生合成の主たる活性を担っている(38,39)。一方SMS2はゴルジ体の他、形質膜に局在しており、TNF-α等の刺激で生成されたセラミドを再びスフィンゴミエリンに戻す、いわゆるスフィンゴミエリンサイクルに関与すると考えられている(39)。SMS1のゴルジ体局在はCOG(Conserved Oligomeric Golgi)複合体に依存しており、Cog2欠損細胞ではSMS1の細胞内局在が変化し、スフィンゴミエリンの合成量が減少する(40)。SMSは6つの膜貫通ドメインを有しており、また触媒活性はゴルジ体内腔もしくは細胞外側に面していることから、セラミドはゴルジ体の内腔側でスフィンゴミエリンに変換されると考えられる(38,41)。哺乳動物細胞ではこれら2つの酵素に加え、SMS-related(SMSr)タンパク分子が存在する。SMSrは小胞体に局在しており、スフィンゴミエリン生合成活性は見られないが、哺乳動物細胞では珍しいセラミドホスホエタノールアミン(CPE)を生成する(42)。ちなみにこのCPE生合成活性はSMS1やSMS2でも見られる(43,44)。SMSr遺伝子のノックアウトマウスは見かけ正常であり、このタンパクの生理的な機能は不明である(44)。
ガラクトシルセラミド(GalCer)合成酵素
GalCer(別名セレブロシド)はUDP-ガラクトース:セラミドガラクトース転移酵素(GalCerS、遺伝子名UGT8)により、UDP-ガラクトースのガラクトースをセラミドの水酸基に転移させることにより産生される(Gal β1–Cer)。基質として脂肪酸のC2位が水酸化されたセラミド(αヒドロキシセラミド)を優先的に使用するが、非ヒドロキシセラミドやジアシルグリセロール(モノガラクトシルジアシルグリセロールを生成)も基質となる(45–47)。なおαヒドロキシセラミドのαヒドロキシ脂肪酸は小胞体に存在する脂肪酸2-水酸化酵素(F2AH)により生成される(48)。GalCerSは主に神経系においてミエリン形成に重要なオリゴデンドロサイトやシュワン細胞、あるいは腎臓や睾丸に発現している。この酵素は糖転移酵素としては珍しくI型の膜貫通タンパク質で、細胞質領域に小胞体保留シグナル(KKVK)を有しているため、主に小胞体に局在しており、活性部位は小胞体内腔側に位置している(49)。ガラクトシルセラミドはさらに硫酸基転移酵素により硫酸化されスルファチド(Gal (3-SO4) β1-Cer)に(50)、GM3合成酵素によりシアロ化されGM4(Sia α2,3 Gal β1-Cer)に(51)、そしてGb3合成酵素によりガラクトシル化されガラビオシルセラミド(Gb2:Gal α1,4 Gal β1-Cer)に(52)、それぞれ変換される。なおこれらGalCer由来糖脂質をガラ系列糖脂質と呼ぶ。
グルコシルセラミド(GlcCer)合成酵素
GlcCer(別名グルコセレブロシド)はGalCer由来糖脂質を除く糖脂質の前駆体であり、UDP-グルコース:セラミドグルコース転移酵素(GlcCerS、遺伝子名UGCG)によりUDP-グルコースからグルコースをセラミドに転移させることで産生される(Glc β1–Cer)(53)。GlcCerSはN末端側に1つ、C末端側に複数の膜貫通領域を有しており、その間を親水性のループで繋いた構造をしている。主にシス/メディアゴルジ槽(及び小胞体)に存在していると考えられ、N末端の膜貫通領域はゴルジ体への局在能を有する(38, 54)。ただし内在性タンパクの正確な局在は不明である。親水性ループの中には活性に必要なモチーフ(D1、D2、D3及び(Q/R)XXRW)が含まれており、またこのループは細胞質に面していることから、糖脂質生合成の中ではGlcCerが唯一細胞質側で生成されると考えられる(55)。GlcCerからは下記の糖脂質が生成される他、グルコセレブロシダーゼGBA1及びGBA2によりGlcCerのグルコースがコレステロールに転移され、コレステリルグルコシドが生成される(56, 57)。
ラクトシルセラミド(LacCer)合成酵素
LacCerはUDP-ガラクトースのガラクトースをGlcCerにβ1,4結合で付加させることにより生成される。このLacCerの生合成反応は2種類の酵素、B4GalT5及びB4GalT6が司る(58, 59)。ノックアウトマウスやノックアウト細胞を用いた研究より、B4GalT5のほうが主要なLacCer合成酵素として機能する(59–63)。LacCer及びそれ以降の糖脂質生合成酵素は共通してII型の膜貫通タンパク構造を有しており、活性部位をゴルジ体(トランスゴルジネットワーク(TGN)を含む)の内腔側に有している(64)。LacCerが生成されるためには、細胞質側で生成されたGlcCerが脂質二重膜を横断(フロップ)し内腔側に輸送されなければいけない。このGlcCerのフロッパーゼ(もしくはスクランブラーゼ)の活性は検出されているものの(65)、分子同定には至っていない。
LacCer以降の糖脂質の生合成酵素
LacCerに様々な糖が付加することで、多様な糖脂質が生成される。詳細は第4章を参照していただくとして、ここでは3糖付加の糖脂質に絞りその概要を述べる。
(1) GM3生合成(ガングリオ系糖脂質の基点)
GM3はGM3合成酵素(GM3S/ST3Gal5)がCMP-シアル酸のシアル酸をLacCerにα2,3結合で付加することにより生成される(66)。GM3Sは主にゴルジ体に局在しており、Brefeldin A処理によりゴルジ槽と共に小胞体に局在が変化することより、TGNでなくゴルジ槽側に局在すると考えられる(38,62)。この細胞内局在はSMS1と同様COG複合体に依存している(40)。このGM3を基点に様々なガングリオシドが生成される。
(2) Gb3Cer(グロボトリアオシルセラミド, CTH:グロボ系糖脂質の基点)
Gb3CerはGb3合成酵素(Gb3S/A4GalT)がUDP-ガラクトースのガラクトースをLacCerにα1,4結合で付加することにより生成される(52)。BrefeldinA処理によりDot状の局在(エンドソームとの融合)が見られること、またBrefeldinA処理をはじめGb3Sの局在を変化させる条件下で、TGNマーカーもGb3Sと同様の局在変化を見せることから、Gb3SはGM3Sとは異なり主にTGNに局在していると考えられる(62,67,68)。
(3) iGb3Cer(イソグロボトリアオシルセラミド:イソグロボ系糖脂質の基点)
iGb3はiGb3合成酵素(A3GalT2)がUDP-ガラクトースのガラクトースをLacCerにα1,3結合で付加することにより生成される(69)。なおヒトでは発現していないためiGb3は生成されない(70)。iGb3は一時期NKT細胞の内在性リガンドと言われていたが(71)、ヒトに発現していないため疑問視されている。
(4) Lc3Cer(アミノCTH:ラクト系及びネオラクト系糖脂質の基点)
Lc3CerはLc3合成酵素(B3GNT5)がUDP-N-アセチルグルコサミン(GlcNAc)のGlcNAcをLacCerにβ1,3結合で付加することにより生成される(72,73)。GM3Sと同様、トランスゴルジ槽の局在と考えられている(67)。ABO血液型糖脂質はこのシリーズにより生成される。
(5) GA2(アシアロGM2; アシアロガングリオ系糖脂質の基点)
GA2はGM2/GA2/GD2合成酵素(B4GALNT1)がUDP-N-アセチルガラクトサミン(UDP-GalNAc)のGalNAcをLacCerにβ1,4結合で付加することにより生成される(74–76)。この酵素は基質がGM3であればGM2を、GD3であればGD2を生成する。同酵素はLacCerを基質とした場合、Vmax/Km比がGM3やGM2の時より低いため(76)、GA2及びアシアロガングリオ系糖脂質はGM3の生合成が抑制されているときに生成されると考えられる。この酵素はGb3Sと同様、主にTGNに存在する(77)。
セラミドキナーゼ(セラミド-1-リン酸生合成)
セラミドの水酸基がリン酸化されるとセラミド-1-リン酸(C1P)となる(78)。C1P産生経路の1つは、セラミドキナーゼ(CERK)によるセラミドのリン酸化により生成されると考えられている(79)。CERKはジアシルグリセロールキナーゼファミリーの一員で、スフィンゴシンキナーゼと類似の構造をしている。この酵素は細胞質に存在しているが、ミリストイル化される配列を持ち、またN末端にpleckstrin homology (PH)ドメインを有することから、膜の細胞質側に結合しC1Pを生合成すると考えられる(79)。実際このPHドメインを介してTGN、エンドソーム、形質膜、ミトコンドリアに結合する(80,81)。驚いたことにCERKノックアウトマウスにおいて脳のC1P量は減少しなかったことより、別のC1P生合成経路が存在すると考えられる(82)。
4.スフィンゴ脂質の膜間輸送
セラミド輸送タンパクCERT
セラミドは主に小胞体で産生され、スフィンゴミエリンやGlcCerはゴルジ体で生合成されることから、セラミドは小胞体からゴルジ体へ何かしらの機構で輸送されなければならない。De novoで生合成されたセラミドは少なくとも2経路よりゴルジ体に輸送される。1つはATP及び細胞質画分依存性の輸送でスフィンゴミエリン生合成のためのセラミド供給の主な経路であり、もう1つはATP及び細胞質画分非依存性の輸送である(83)。セラミド輸送タンパク(CERT)による輸送は前者の輸送を司る。CERTの詳細は第18章を参考にしていただくとして、ここでは簡単な機能を記す。CERTは細胞質に存在する可溶性タンパクで、C末端側のsteroidgenic acute regulatory protein-related lipid transfer (START)ドメインによりATP非依存的にセラミドの膜間輸送を行う(84,85)。少ない発現量でセラミドを小胞体からゴルジ体へと効率的に輸送するため、CERTは膜と結合する2つの領域を有する。1つ目はN末端側のPHドメインで、主にゴルジ体(特にトランスゴルジ側)に存在しているPI4Pと結合する(5)。2つ目は中間部位のtwo phenylalanines in an acidic tract(FAAT)モチーフで、小胞体に存在している膜タンパクVAP-A及びVAP-Bと結合する(86)。CERT経路がATP依存性であるのは恐らくPI4Pの生成にエネルギーが使用されるためと推測される。CERTを欠損した細胞においてスフィンゴミエリンの生合成は30%前後に減少するが、GlcCerの生合成には影響しないことより、CERTはSMSの局在するトランスゴルジ槽に選択的に輸送していると考えられる(5,63)。
一方スフィンゴミエリン生合成におけるセラミドのCERT非依存経路(マイナー経路)及びGlcCerに使用されるセラミドのCERT非依存経路は解明されていない。GlcCerを含め糖脂質のセラミドはスフィンゴミエリンと比較しC24:0やC24:1の極長鎖脂肪酸セラミドが優先的に利用されていることから、GlcCerへのセラミド輸送にセラミドの脂肪酸の鎖長が関与している可能性がある(87,88)。
GlcCer輸送タンパクFAPP2
GlcCerは主にシス/ミディアゴルジの細胞質側で生合成され、その後内腔側へとフロップされるが、その前に膜間輸送が行われる場合がある。糖脂質の膜間輸送を行うタンパクとしてGlycolipid transfer protein(GLTP)ファミリーが知られているが(89)、そのファミリーの一員であるFour-phosphate adaptor protein 2(FAPP2)はC末側のGLTPドメインを介してGlcCerの非小胞性膜間輸送を司り、GlcCer以降の糖脂質生合成の代謝に影響を及ぼす(62,90)。FAPP2は細胞質に存在する可溶性タンパクで、N末側にPI4P及びArf1(主にTGNに局在)と結合するPHドメインを有しているため、シスゴルジ槽で生成されたGlcCerをTGNに直接輸送すると考えられている。それはFAPP2を遺伝子ノックダウンあるいはノックアウトすると、TGN局在酵素によって生合成されるGb3CerやGA2は減少するが、酵素がゴルジ槽側に存在するGM3は減少しないことからも支持される(62)。一方FAPP2がGlcCerをゴルジ体から小胞体に逆輸送するとの報告もあり(38)、機能に関して更なる解析が必要である。
C1P輸送タンパクCPTP
細胞質側で生成されたC1PはC1P transfer protein(CPTP)により膜間輸送され得る(91)。CPTPはGLTP様のドメインを有しており、FAPP2と同様GLTPファミリーに属するが、基質として糖脂質は輸送せずC1Pを輸送する。CPTPは細胞質のタンパクであるが、PHドメイン等明らかな膜結合ドメインを持っていないため、C1Pがどこからどこに膜間輸送されるかは更なる解析が必要である。CPTPを発現抑制した細胞ではTGNやエンドソーム等におけるC1P量が増加し、形質膜のC1P量が減少することにより、TGNから形質膜に輸送されていると推測される(91)。
5.おわりに
スフィンゴ脂質生合成の概要について述べてきたが、スフィンゴ脂質は頭部の多様性はもちろんのこと、脂質部位に関してもスフィンゴ塩基及び脂肪酸において多様性を有しており、その生合成機構もかなり解明されてきた。これら生合成酵素の発現制御はスフィンゴ脂質の様々な病態への関与を考える上で今後益々重要になるであろう。一方スフィンゴ脂質の生合成が理路整然と行われるためには、生合成酵素の細胞内局在(輸送)、及び基質輸送の両者が正しく制御されていなければならない。生合成酵素の多くは発現量が少なくとも機能しており、細胞は少ない酵素を効率よく使用出来るよう様々な制御機構を備えていると考えられる。COG複合体等、一般的なゴルジ体の形成・輸送に関する因子の他、特異的に制御するタンパクも存在していると考えられ、今後の研究が期待される。
付記
本コンテンツは、セラミド研究会が編集した『セラミド研究の新展開~基礎から応用へ~』(発行日,2019年6月1日;発行者,株式会社・食品化学新聞社)に上梓した総説を一部内容改変して本研究会Webサイトに転載するものです。本コンテンツの著作権は食品化学新聞社に帰属しており、セラミド研究会は転載することの承認を食品化学新聞社から得ています。
本コンテンツは、クリエイティブ・コモンズ の定めたCC BY 4.0ライセンスの条件で掲載しており、著者と著作権所有者が明記され、かつ、日本セラミド研究会からの出版物である旨が引用されていることを条件として、他の会での使用、配布、または複製は許可されています。これらの条件に準拠していない使用,配布,または複製は許可されていません。
CC BY 4.0ライセンスの内容については以下URLを参照してください。
https://creativecommons.org/licenses/by/4.0/
・「図2 スフィンゴ脂質生合成経路」は Trafficの総説の図を一部改変した(92)。
引用文献
- Hanada K, et al.: Sphingolipids are essential for the growth of Chinese hamster ovary cells. Restoration of the growth of a mutant defective in sphingoid base biosynthesis by exogenous sphingolipids. J Biol Chem, 267, 23527-23533 (1992)
- Pinto WJ, et al.: Characterization of enzymatic synthesis of sphingolipid long-chain bases in Saccharomyces cerevisiae: mutant strains exhibiting long-chain-base auxotrophy are deficient in serine palmitoyltransferase activity. J Bacteriol, 174, 2575-2581 (1992)
- Adachi-Yamada T, et al.: De novo synthesis of sphingolipids is required for cell survival by down-regulating c-Jun N-terminal kinase in Drosophila imaginal discs. Mol Cell Biol, 19, 7276-7286 (1999)
- Merrill AH Jr.: Characterization of serine palmitoyltransferase activity in Chinese hamster ovary cells. Biochim Biophys Acta, 754, 284-291 (1983)
- Nagiec MM, et al.: The LCB2 gene of Saccharomyces and the related LCB1 gene encode subunits of serine palmitoyltransferase, the initial enzyme in sphingolipid synthesis. Proc Natl Acad Sci USA, 91, 7899-7902 (1994)
- Weiss B, Stoffel W.: Human and murine serine-palmitoyl-CoA transferase–cloning, expression and characterization of the key enzyme in sphingolipid synthesis. Eur J Biochem, 249, 239-247 (1997)
- Hanada K, et al.: A mammalian homolog of the yeast LCB1 encodes a component of serine palmitoyltransferase, the enzyme catalyzing the first step in sphingolipid synthesis. J Biol Chem, 272, 32108-32114 (1997)
- Hanada K, et al.: Purification of the serine palmitoyltransferase complex responsible for sphingoid base synthesis by using affinity peptide chromatography techniques. J Biol Chem, 275, 8409-8415 (2000)
- Hornemann T, et al.: Cloning and initial characterization of a new subunit for mammalian serine-palmitoyltransferase. J Biol Chem, 281, 37275-37281 (2006)
- Han G, et al.: Identification of small subunits of mammalian serine palmitoyltransferase that confer distinct acyl-CoA substrate specificities. Proc Natl Acad Sci USA, 106, 8186-8191 (2009)
- Hornemann T, et al.: The SPTLC3 subunit of serine palmitoyltransferase generates short chain sphingoid bases. J Biol Chem, 284, 26322-26330 (2009)
- Gable K, et al.: A disease-causing mutation in the active site of serine palmitoyltransferase causes catalytic promiscuity. J Biol Chem, 285, 22846-22852 (2010)
- Esaki K, et al.: L-Serine Deficiency Elicits Intracellular Accumulation of Cytotoxic Deoxysphingolipids and Lipid Body Formation. J Biol Chem, 290, 14595-14609 (2015)
- Mandon EC, et al.: Subcellular localization and membrane topology of serine palmitoyltransferase, 3-dehydrosphinganine reductase, and sphinganine N-acyltransferase in mouse liver. J Biol Chem, 267:11144-11148 (1992)
- Yasuda S, et al.: Localization, topology, and function of the LCB1 subunit of serine palmitoyltransferase in mammalian cells. J Biol Chem, 278, 4176-4183 (2003)
- Beeler T, et al.: The Saccharomyces cerevisiae TSC10/YBR265w gene encoding 3-ketosphinganine reductase is identified in a screen for temperature-sensitive suppressors of the Ca2+-sensitive csg2Delta mutant. J Biol Chem, 273, 30688-30694 (1998)
- Kihara A, Igarashi Y.: FVT-1 is a mammalian 3-ketodihydrosphingosine reductase with an active site that faces the cytosolic side of the endoplasmic reticulum membrane. J Biol Chem, 279, 49243-49250 (2004)
- Pewzner-Jung Y, et al.: When do Lasses (longevity assurance genes) become CerS (ceramide synthases)?: Insights into the regulation of ceramide synthesis. J Biol Chem, 281, 25001-25005 (2006)
- Levy M, Futerman: Mammalian ceramide synthases. IUBMB Life, 62, 347-356 (2010)
- Venkataraman K, et al.: Upstream of growth and differentiation factor 1 (uog1), a mammalian homolog of the yeast longevity assurance gene 1 (LAG1), regulates N-stearoyl-sphinganine (C18-(dihydro)ceramide) synthesis in a fumonisin B1-independent manner in mammalian cells. J Biol Chem, 277, 35642-35649 (2002)
- Riebeling C, et al.: Two mammalian longevity assurance gene (LAG1) family members, trh1 and trh4, regulate dihydroceramide synthesis using different fatty acyl-CoA donors. J Biol Chem, 278, 43452-43459 (2003)
- Laviad EL, et al.: Characterization of ceramide synthase 2: tissue distribution, substrate specificity, and inhibition by sphingosine 1-phosphate. J Biol Chem, 283, 5677-5684 (2008)
- Mizutani Y, et al.: Mammalian Lass6 and its related family members regulate synthesis of specific ceramides. Biochem J, 390, 263-271 (2005)
- Mizutani Y, et al.: LASS3 (longevity assurance homologue 3) is a mainly testis-specific (dihydro)ceramide synthase with relatively broad substrate specificity. Biochem J, 398, 531-538 (2006)
- Hirschberg K, et al.: The long-chain sphingoid base of sphingolipids is acylated at the cytosolic surface of the endoplasmic reticulum in rat liver. Biochem J, 290, 751-757 (1993)
- Tidhar R, et al.: Eleven residues determine the acyl chain specificity of ceramide synthases. J Biol Chem, 293, 9912-9921 (2018)
- Ternes P, et al.: Identification and characterization of a sphingolipid delta 4-desaturase family. J Biol Chem, 277, 25512-25518 (2002)
- Omae F, et al.: DES2 protein is responsible for phytoceramide biosynthesis in the mouse small intestine. Biochem J, 379, 687-695 (2004)
- Endo K, et al.: Degenerative spermatocyte, a novel gene encoding a transmembrane protein required for the initiation of meiosis in Drosophila spermatogenesis. Mol Gen Genet, 253, 157-65 (1996)
- Beauchamp E, et al.: Myristic acid increases the activity of dihydroceramide Delta4-desaturase 1 through its N-terminal myristoylation. Biochimie, 89, 1553-1561 (2007)
- Cadena DL, et al.: The product of the MLD gene is a member of the membrane fatty acid desaturase family: overexpression of MLD inhibits EGF receptor biosynthesis. Biochemistry, 36, 6960-6967 (1997)
- Michel C, et al.: Characterization of ceramide synthesis. A dihydroceramide desaturase introduces the 4,5-trans-double bond of sphingosine at the level of dihydroceramide. J Biol Chem, 272, 22432-22437 (1997)
- Michel C, et al.: Conversion of dihydroceramide to ceramide occurs at the cytosolic face of the endoplasmic reticulum. FEBS Lett, 416, 153-155 (1997)
- Enomoto A, et al.: Dihydroceramide:sphinganine C-4-hydroxylation requires Des2 hydroxylase and the membrane form of cytochrome b5. Biochem J, 397, 289-295 (2006)
- Marggraf WD, et al.: The formation of sphingomyelin from phosphatidylcholine in plasma membrane preparations from mouse fibroblasts. Biochim Biophys Acta, 664, 61-73 (1981)
- Huitema K, et al.: Identification of a family of animal sphingomyelin synthases. EMBO J, 23, 33-44 (2004)
- Yamaoka S, et al.: Expression cloning of a human cDNA restoring sphingomyelin synthesis and cell growth in sphingomyelin synthase-defective lymphoid cells. J Biol Chem, 279, 18688-18693 (2004)
- Halter D, et al.: Pre- and post-Golgi translocation of glucosylceramide in glycosphingolipid synthesis. J Cell Biol, 179, 101-115 (2007)
- Tafesse FG, et al.: Both sphingomyelin synthases SMS1 and SMS2 are required for sphingomyelin homeostasis and growth in human HeLa cells. J Biol Chem, 282, 17537-17547 (2007)
- Spessott W, et al.: Cog2 null mutant CHO cells show defective sphingomyelin synthesis. J Biol Chem, 285, 41472-41482 (2010)
- Futerman AH, et al.: Sphingomyelin synthesis in rat liver occurs predominantly at the cis and medial cisternae of the Golgi apparatus. J Biol Chem, 265, 8650-8657 (1990)
- Vacaru AM, et al.: Sphingomyelin synthase-related protein SMSr controls ceramide homeostasis in the ER. J Cell Biol, 185, 1013-1027 (2009)
- Ternes P, et al.: Sphingomyelin synthase SMS2 displays dual activity as ceramide phosphoethanolamine synthase. J Lipid Res, 50, 2270-2277 (2009)
- Ding T, et al.: All members in the sphingomyelin synthase gene family have ceramide phosphoethanolamine synthase activity. J Lipid Res, 56, 537-545 (2015)
- Schulte S, Stoffel W.: Ceramide UDPgalactosyltransferase from myelinating rat brain: purification, cloning, and expression. Proc Natl Acad Sci USA, 90, 10265-10269 (1993)
- Schaeren-Wiemers N, et al.: The UDP-galactose:ceramide galactosyltransferase: expression pattern in oligodendrocytes and Schwann cells during myelination and substrate preference for hydroxyceramide. J Neurochem, 65, 2267-2278 (1995)
- van der Bijl P, et al.: Synthesis of non-hydroxy-galactosylceramides and galactosyldiglycerides by hydroxy-ceramide galactosyltransferase. Biochem J, 317, 589-597 (1996)
- Alderson NL, et al.: The human FA2H gene encodes a fatty acid 2-hydroxylase. J Biol Chem, 279, 48562-48568 (2004)
- Sprong H, et al.: UDP-galactose:ceramide galactosyltransferase is a class I integral membrane protein of the endoplasmic reticulum. J Biol Chem, 273, 25880-25888 (1998)
- Honke K, et al.: Molecular cloning and expression of cDNA encoding human 3′-phosphoadenylylsulfate:galactosylceramide 3′-sulfotransferase. J Biol Chem, 272, 4864-8 (1997)
- Chisada S, et al.: Zebrafish and mouse alpha2,3-sialyltransferases responsible for synthesizing GM4 ganglioside. J Biol Chem, 284, 30534-46 (2009)
- Okajima T, et al.: Molecular cloning of globotriaosylceramide/CD77 synthase, a glycosyltransferase that initiates the synthesis of globo series glycosphingolipids. J Biol Chem, 275, 40498-503 (2000)
- Ichikawa S, et al.: Expression cloning of a cDNA for human ceramide glucosyltransferase that catalyzes the first glycosylation step of glycosphingolipid synthesis. Proc Natl Acad Sci USA, 93, 4638-4643 (1996)
- Kohyama-Koganeya A, et al.: Drosophila glucosylceramide synthase: a negative regulator of cell death mediated by proapoptotic factors. J Biol Chem, 279, 35995-36002 (2004)
- Marks DL, et al.: Identification of active site residues in glucosylceramide synthase. A nucleotide-binding catalytic motif conserved with processive beta-glycosyltransferases. J Biol Chem, 276, 26492-26498 (2001)
- Akiyama H, et al.: Cholesterol glucosylation is catalyzed by transglucosylation reaction of β-glucosidase 1. Biochem Biophys Res Commun, 441, 838-43 (2013)
- Marques AR, et al.: Glucosylated cholesterol in mammalian cells and tissues: formation and degradation by multiple cellular β-glucosidases. J Lipid Res, 57, 451-63 (2016)
- Nomura T, et al.: Purification, cDNA cloning, and expression of UDP-Gal: glucosylceramide beta-1,4-galactosyltransferase from rat brain. J Biol Chem, 273, 13570-13577 (1998)
- Kumagai T, et al.: Involvement of murine β-1,4-galactosyltransferase V in lactosylceramide biosynthesis. Glycoconj J, 27, 685-695 (2010)
- Nishie T, et al.: Beta4-galactosyltransferase-5 is a lactosylceramide synthase essential for mouse extra-embryonic development. Glycobiology, 20, 1311-1322 (2010)
- Tokuda N, et al.: β4GalT6 is involved in the synthesis of lactosylceramide with less intensity than β4GalT5. Glycobiology, 23, 1175-1183 (2013)
- D’Angelo G, et al.: Vesicular and non-vesicular transport feed distinct glycosylation pathways in the Golgi. Nature, 501, 116-120 (2013)
- Yamaji T, Hanada K.: Establishment of HeLa cell mutants deficient in sphingolipid-related genes using TALENs. PLoS One, 9, e88124 (2014)
- Lannert H, et al.: Lactosylceramide is synthesized in the lumen of the Golgi apparatus. FEBS Lett 342, 91-96 (1994)
- Chalat M, et al.: Reconstitution of glucosylceramide flip-flop across endoplasmic reticulum: implications for mechanism of glycosphingolipid biosynthesis. J Biol Chem, 287, 15523-15532 (2012)
- Ishii A, et al.: Expression cloning and functional characterization of human cDNA for ganglioside GM3 J Biol Chem, 273, 31652-31655 (1998)
- Sherwood AL, Holmes EH.: Brefeldin A induced inhibition of de novo globo- and neolacto-series glycolipid core chain biosynthesis in human cells. Evidence for an effect on beta 1–>4galactosyltransferase activity. J Biol Chem, 267, 25328-25336 (1992)
- Yamaji T, et al.: Transmembrane BAX inhibitor motif containing (TMBIM) family proteins perturbs a trans-Golgi network enzyme, Gb3 synthase, and reduces Gb3 biosynthesis. J Biol Chem, 285, 35505-35518 (2010)
- Keusch JJ, et al.: Expression cloning of a new member of the ABO blood group glycosyltransferases, iGb3 synthase, that directs the synthesis of isoglobo-glycosphingolipids. J Biol Chem, 275, 25308-25314 (2000)
- Christiansen D, et al.: Humans lack iGb3 due to the absence of functional iGb3-synthase: implications for NKT cell development and transplantation. PLoS Biol, 15, e172 (2008)
- Zhou D, et al.: Lysosomal glycosphingolipid recognition by NKT cells. Science, 306, 1786-1789 (2004).
- Henion TR, et al.: Cloning of a mouse beta 1,3 N-acetylglucosaminyltransferase GlcNAc(beta 1,3)Gal(beta 1,4)Glc-ceramide synthase gene encoding the key regulator of lacto-series glycolipid biosynthesis. J Biol Chem, 276, 30261-30269 (2001)
- Togayachi A, et al.: Molecular cloning and characterization of UDP-GlcNAc:lactosylceramide beta 1,3-N-acetylglucosaminyltransferase (beta 3Gn-T5), an essential enzyme for the expression of HNK-1 and Lewis X epitopes on glycolipids. J Biol Chem, 276, 22032-22040 (2001)
- Nagata Y, et al.: Expression cloning of beta 1,4 N-acetylgalactosaminyltransferase cDNAs that determine the expression of GM2 and GD2 gangliosides. J Biol Chem, 267, 12082-12089 (1992)
- Hidari JK, et al.: beta 1-4N-acetylgalactosaminyltransferase can synthesize both asialoglycosphingolipid GM2 and glycosphingolipid GM2 in vitro and in vivo: isolation and characterization of a beta 1-4N-acetylgalactosaminyltransferase cDNA clone from rat ascites hepatoma cell line AH7974F. Biochem J, 303, 957-965 (1994)
- Yamashiro S, et al.: Substrate specificity of beta 1,4-N-acetylgalactosaminyltransferase in vitro and in cDNA-transfected cells. GM2/GD2 synthase efficiently generates asialo-GM2 in certain cells. J Biol Chem, 27, 6149-6155 (1995)
- Giraudo CG, et al.: GA2/GM2/GD2 synthase localizes to the trans-golgi network of CHO-K1 cells. Biochem J, 342, 633-640 (1999)
- Bajjalieh SM, et al.: Synaptic vesicle ceramide kinase. A calcium-stimulated lipid kinase that co-purifies with brain synaptic vesicles. J Biol Chem, 264, 14354-14360 (1989)
- Sugiura M, et al.: Ceramide kinase, a novel lipid kinase. Molecular cloning and functional characterization. J Biol Chem, 277, 23294-23300 (2002)
- Carré A, et al.: Ceramide kinase targeting and activity determined by its N-terminal pleckstrin homology domain. Biochem Biophys Res Commun, 324, 1215-1219 (2004)
- Kim TJ, et al.: The interaction between the pleckstrin homology domain of ceramide kinase and phosphatidylinositol 4,5-bisphosphate regulates the plasma membrane targeting and ceramide 1-phosphate levels. Biochem Biophys Res Commun, 342, 611-617 (2006)
- Mitsutake S, et al.: The generation and behavioral analysis of ceramide kinase-null mice, indicating a function in cerebellar Purkinje cells. Biochem Biophys Res Commun, 363, 519-524 (2007)
- Fukasawa M, et al.: Genetic evidence for ATP-dependent endoplasmic reticulum-to-Golgi apparatus trafficking of ceramide for sphingomyelin synthesis in Chinese hamster ovary cells. J Cell Biol, 144, 673-685 (1999)
- Hanada K, et al.: Molecular machinery for non-vesicular trafficking of ceramide. Nature, 2003;426:803-809.
- Hanada K.: Co-evolution of sphingomyelin and the ceramide transport protein CERT. Biochim Biophys Acta, 1841, 704-719 (2014)
- Kawano M, et al.: Efficient trafficking of ceramide from the endoplasmic reticulum to the Golgi apparatus requires a VAMP-associated protein-interacting FFAT motif of CERT. J Biol Chem, 281, 30279-30288 (2006)
- Loizides-Mangold U, et al.: Glycosylphosphatidylinositol anchors regulate glycosphingolipid levels. J Lipid Res, 53, 1522-1534 (2012)
- Yamaji T, et al.: Role of Intracellular Lipid Logistics in the Preferential Usage of Very Long Chain-Ceramides in Glucosylceramide. Int J Mol Sci, 17, E1761 (2016)
- Mattjus P. Specificity of the mammalian glycolipid transfer proteins. Chem Phys Lipids, 194, 72-78 (2016)
- D’Angelo G, et al.: Glycosphingolipid synthesis requires FAPP2 transfer of glucosylceramide. Nature, 449, 62-67 (2007)
- Simanshu DK, et al.: Non-vesicular trafficking by a ceramide-1-phosphate transfer protein regulates eicosanoids. Nature, 500, 463-467 (2013)
- Yamaji T, Hanada: Sphingolipid metabolism and interorganellar transport: localization of sphingolipid enzymes and lipid transfer proteins. Traffic, 16, 101-122 (2015)